参考文献
- Lu M., Uchil P.D., Li W., Zheng D., Terry D.S., Gorman J., Shi W., Zhang B., Zhou T., Ding S., et al. Real-time conformational dynamics of SARS-CoV-2 spikes on virus particles. Cell Host & Microbe. 2020;28:880–891.e888
- Myong S, Bruno MM, Pyle AM, Ha Spring-loaded mechanism of DNA unwinding by hepatitis C virus NS3 helicase. Science. 2007, 317(5837): 513-516.
- Lee SJ, Syed S, Enemark EJ, Schuck S, Stenlund A, Ha T, Joshua-Tor L. Dynamic look at DNA unwinding by a replicative helicase. Proceedings of the National Academy of Sciences of the United States of America. 2014, 111(9): E827-35.
- Park J, Myong S, Niedziela-Majka A, Lee KS, Yu J, Lohman TM, Ha PcrA helicase dismantles RecA filaments by reeling in DNA in uniform steps. Cell. 2010, 142(4): 544-555.
- Syed S, Pandey M, Patel SS, Ha Single-molecule fluorescence reveals the unwinding stepping mechanism of replicative helicase. Cell Reports. 2014, 6(6): 1037-1045.
- Lakadamyali M, Rust MJ, Babcock HP, et al. Visualizing infection of individual influenza viruses. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100: 9280-9285
- Qin C, Li W, Li Q, et al. Real-time dissection of dynamic uncoating of individual influenza viruses. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116: 2577-2582
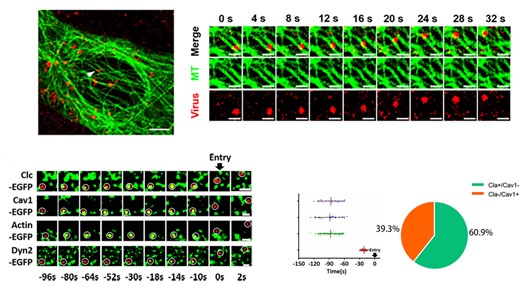
- Li Y, Wang J, Hou W, et al. Dynamic Dissection of the Endocytosis of Porcine Epidemic Diarrhea Coronavirus Cooperatively Mediated by Clathrin and Caveolae as Visualized by Single-Virus Tracking. mBio, 2021, 12: e00256-21